MGI-DNA建库试剂盒
产品介绍
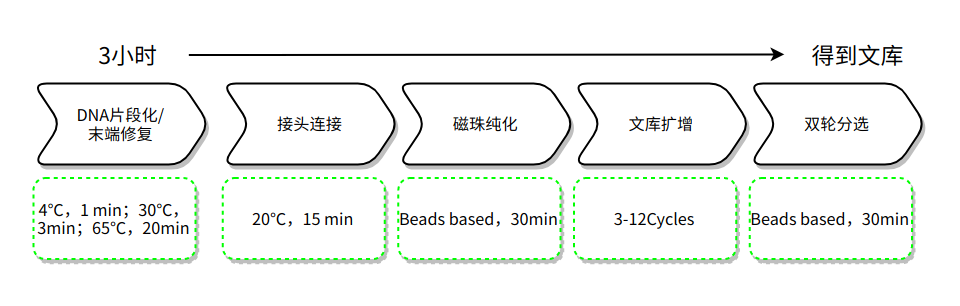
MGI-DNA建库试剂盒是针对MGI®高通量测序平台专业开发设计的新一代酶切法建库试剂盒。与传统的建库法比较,本品采用高质量的片段化酶,摆脱了繁琐的超声过程,同时简化了操作流程,将片段化模块与末端修复模块合二为一,极大的降低了建库的时间和成本。本试剂盒具有优秀的文库转化率,可应用于常规动植物基因组、微生物基因组等样本,同时能兼容FFPE DNA样本的建库。该试剂盒使用了最新优化的连接酶,改善了接头连接时的片段自连现象,同时替换了新型高保真酶,进一步提升了扩增的均一性和保真性。
产品优势
① 适用500 pg-1 μg的基因组DNA、全长cDNA等样本
② 高质量片段化酶,可随机切割双链DNA,酶切片段无偏好性
③ 片段化、末端修复/加A一步完成
④ 强扩增效率的高保真酶,显著提高文库质量及产量
⑤ 适用于FFPE DNA样本
⑥ 严格的批次性能与稳定性质控
产品组分
|
名称 |
96T |
|
Smearase Buffer |
96μL |
|
Smearase Enzyme |
480μL |
|
Ligation Enhancer |
3×960 μL |
|
Novel T4 DNA Ligase |
480μL |
|
Canace Pro Amplification Mix |
3×800 μL |
| Primer Mix for MGI |
480μL |
注意事项
1.关于运输与保存方法:干冰运输。所有组分-20°C保存,有效期1年。
2.关于操作:
■ 为了您的安全和健康,请穿实验服并戴一次性手套操作。
■ 请于使用前将试剂盒各组分置于室温解冻。解冻后上下颠倒数次充分混匀,短暂离心后置于冰上待用。
■ 配制各步骤反应液时推荐使用移液器吹打混匀或轻轻振荡,剧烈振荡可能会造成文库产出下降。
■ 为避免样品交叉污染,推荐使用带滤芯的枪头,吸取不同样品时请更换枪头。
■ 推荐在带热盖的PCR仪中进行各步骤反应,使用前应预热PCR仪至反应温度附近。
■ PCR产物因操作不当极容易产生气溶胶污染,进而影响实验结果准确性。推荐将PCR反应体系配制区和PCR产物纯化检测区进行强制性的物理隔离;使用专用的移液器等设备;并定时对各实验区域进行清洁(使用0.5%次氯酸钠或10%漂白剂进行擦拭清理),以保证实验环境的洁净度。
■ 本产品仅作科研用途!
3.关于DNA片段化
■ 本试剂盒兼容范围为500 pg-1 μg Input DNA。应尽可能使用A260/A280 = 1.8-2.0的高质量Input DNA。
■ 若Input DNA中引入高浓度金属离子螯合剂或其他盐,可能会影响后续实验,建议将DNA稀释在TE(10 mM Tris-HCl ,1 mM EDTA,pH 8.0)中进行片段化。
■ 对于常规的高质量基因组DNA,酶切时间参考表5,本试剂盒片段化偏好低,耐受各种GC含量的模板。对于不同降解程度的FFPE样本,酶切时间参考表6。以上为推荐时间,需客户在自己的实验体系中进行微调,以达到最佳效果。
■ 为保证优质精确的片段化效果,片段化反应配制过程请于冰上操作。
■ 针对FFPE DNA建库,若样本质量不佳,客户对当前建库产量不满意,可选购我公司的FFPE DNA Repair Reagent 对FFPE DNA进行修复,具体使用方法可参考此产品说明书。该试剂可与片段化末修加A过程同时进行,无需额外的操作。
4.关于接头连接(Adapter Ligation)
■ 试剂盒标配接头:th Complete Adaper Kit for MGI。
■ Adapter的质量和使用浓度直接影响连接效率及文库产量。请按照表1和实际DNA投入量确实确定接头用量,需要稀释接头时,请用TE buffer对接头进行稀释。
表1.500pg-1 μg Input DNA推荐的Adapter使用量
|
Input DNA |
10μM Adapter稀释倍数 |
稀释后投入量(μL) |
|
31ng-1 μg |
不稀释 |
5 |
|
11-30 ng |
5 |
5 |
|
3-10 ng |
10 |
5 |
|
0.5-2 ng |
20 |
5 |
5.关于磁珠纯化与分选
■ DNA片段长度分选步骤可选择在末端修复/dA尾添加之前,或接头连接后,或文库扩增后进行。
■ 当Input DNA质量≥50 ng,您可选择在接头连接后分选;如Input DNA质量<50 ng,建议您在文库扩增后进行分选。
■ Ligation Enhancer中包含高浓度的PEG,会对双轮分选产生显著影响。因此,如在接头连接后进行长度分选,必须先进行纯化步骤,再进行双轮分选步骤;如在末端修复/dA尾添加之前或文库扩增后进行长度分选,可直接进行双轮磁珠分选步骤。
■ 磁珠使用前应先平衡至室温,否则会导致得率下降、分选效果不佳。
■ 磁珠每次使用前都应充分振荡混匀或使用移液器上下吹打充分混匀。
■ 转移上清时,请勿吸取磁珠,即使微量残留都将影响后续文库质量。
■ 磁珠漂洗使用的80%乙醇应现用现配,否则将影响回收效率。
■ 进行长度分选时,初始样品体积应尽量≥100 μL,不足时请用超纯水补齐。以防因样品体积太小导致移液误差增大。
■ 产物洗脱前应将磁珠置于室温干燥。干燥不充分容易造成无水乙醇残留影响后续反应;过分干燥又会导致磁珠开裂进而降低纯化得率。通常情况下,室温干燥3-5 min足以让磁珠充分干燥。
■ DNA纯化或长度分选产物如需保存,可使用TE Buffer洗脱,产物可于4°C可保存1-2周,-20°C可保存1个月。
6.关于文库扩增
文库扩增步骤需要严格控制扩增循环数。循环数不足,将导致文库产量低;循环数过多,又将导致文库偏好性增加、重复度增加、嵌合产物增加、扩增突变积累等多种不良后果。表2列举了使用本试剂盒,获得1 μg文库的推荐循环数。
表2. 500 pg-1 μg Input DNA获得1 μg产物扩增循环数推荐表
|
Input DNA(ng) |
Number of cycles required to generate |
|
1 μg |
|
|
1 μg |
44625 |
|
500 ng |
44657 |
|
200 ng |
44688 |
|
50 ng |
44783 |
|
10 ng |
44846 |
|
1 ng |
13 - 15 |
|
500pg |
14 - 16 |
【注】:建库过程中若进行片段分选,扩增时请参照较高循环数扩增。
7.关于文库质检
■ 通常情况下,构建好的文库可通过长度分布检测和浓度检测来进行质量评价。
■ 文库浓度检测可使用:基于双链DNA荧光染料的方法,如Qubit®、PicoGreen®等;基于qPCR绝对定量的方法。
■ 文库浓度检测不可使用:基于光谱检测的方法,如NanoDrop®等。
■ 推荐使用qPCR方法进行文库浓度检测:Qubit®、PicoGreen®等基于双链DNA荧光染料的浓度测定方法,无法有效区分单端连接Adapter的产物、两端均未连接Adapter的产物以及其他不完整双链结构产物;qPCR绝对定量基于PCR扩增原理,仅定量样品中两端Adapter完整的文库(即可测序的文库),可排除单端或双端都不连接Adapter的不可测序文库干扰。
■ 文库长度分布检测,可通过Agilent Bioanalyzer 2100等基于毛细管电泳或微控流原理的设备进行检测。
操作流程