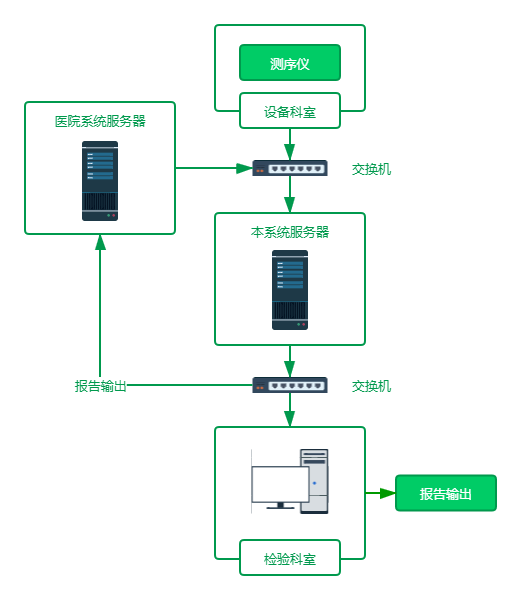
本软件是基于Linux平台研发的应用于计算服务器的软件系统,本软件项目是SaaS[ 是Software-as-a-Service缩写名称,意思为软件即服务,即通过网络提供软件服务。]形式提供的平台软件,客户端只需要浏览器即可,无需安装其他软件。
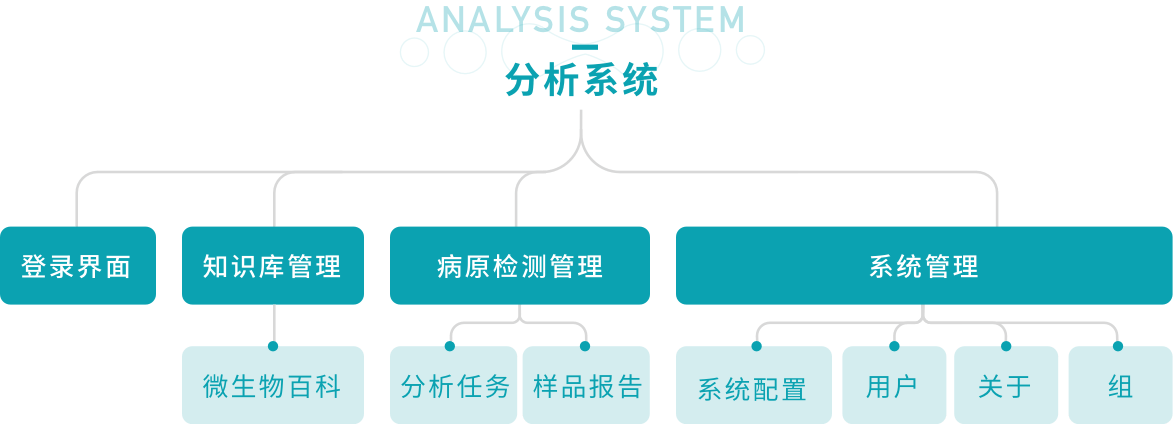
分析软件
| 标号 | 界面描述 | 说 明 |
| 1 | 用户登录界面 | 独立与首页之外的界面,用户必须登录才能查看到首页上的信息,以此来增加系统的安全性。 |
| 2 | 微生物百科 | 管理微生物相关知识,配置控制报告物种检出参数的界面。 |
| 3 | 分析任务 | 按要求填写Excel,并在此界面上传以启动分析,同时可在此界面查看分析状态。 |
| 4 | 样品报告 | 分析完成后,在此处进行检出物种筛选查看与报告发布的界面。 |
| 5 | 系统配置 | 此界面用于病原报告一些参数的配置。 |
| 6 | 用户 | 用户添加,编辑和删除的界面。 |
| 7 | 组 | 组添加,编辑和删除的界面。 |
| 8 | 关于 | 本软件系统信息说明界面。 |
功能描述
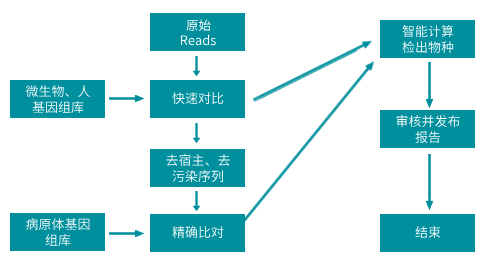
微生物序列参考库模块功能描述
微生物序列参考库模块,用于对已测序微生物基因组序列构建临床关注病原菌参考库和已测序基因组参考库,其中关键步骤为建立序列索引。所述临床关注病原菌参考库和已测序基因组参考库用于样品测序比对分析。
分析流程模块
用于对待检样品测序后的reads采用精确比对到病原菌参考库和快速比对到测序基因参考库,并输出分析结果至交互式Web应用模块。
交互式Web应用模块
用于采用智能算法计算分析结果获取检出物种的列表,通过人工审核在Web应用系统上发布疑似病原菌并生成检测报告,提供报告下载或通过API接口发送报告数据给第三方系统
外部接口
用户接口(用户界面)的类型: Edge,Google,Firefox浏览器
数据接口:基因测序仪下机文件类型为.Fastq格式
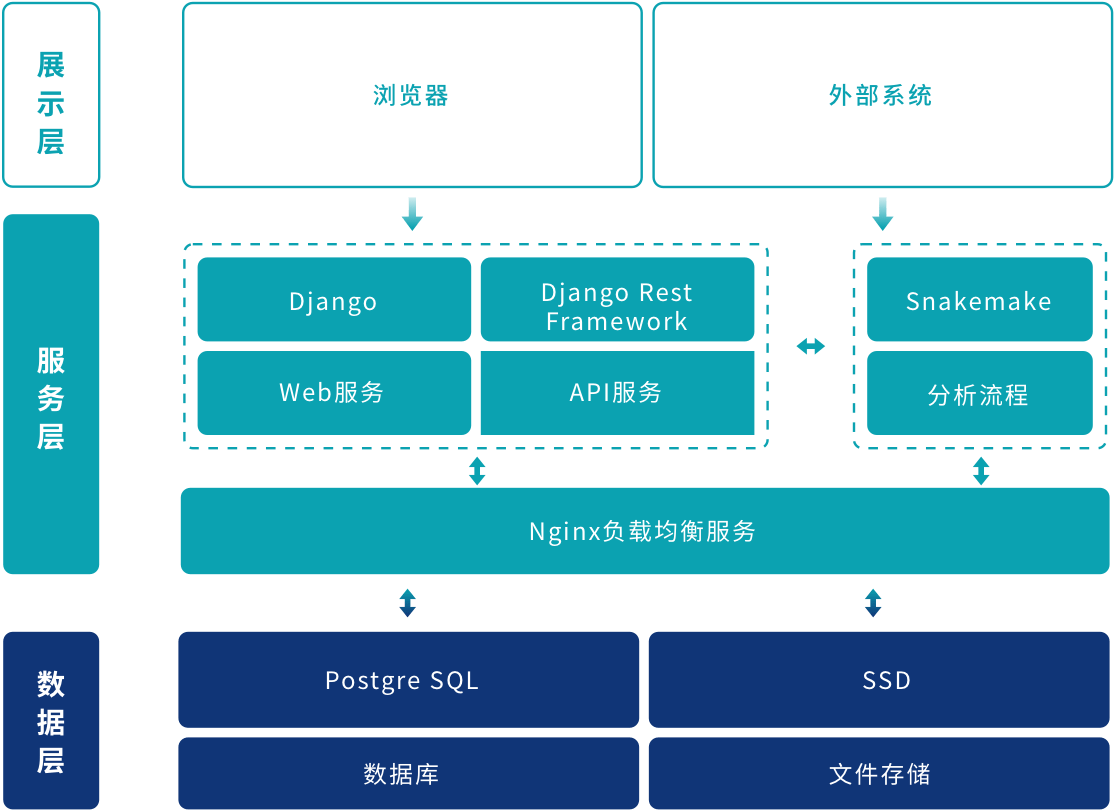
软件体系结构如图所示,分为:展示层(客户端浏览器、外部系统(如:医院HIS系统))、服务层(基于Django框架Web服务和Snakemake分析流程的Nginx负载均衡[ 高性能的HTTP和反向代理web服务器]服务架构)、数据层(PostgreSQL数据库、SSD文件储存)。
| 标号 | 层描述 | 说 明 |
| 1 | 展示层 | 软件是B/S 架构,展示层是基于浏览器的网页形式, |
| 支持Edge,Google,Firefox浏览器。 | ||
| 2 | 服务层 | 是业务逻辑层,包括访问控制相关的系统用户管理、角色管理、 |
| 授权、鉴权、访问日志记录等功能;还包括样本检测查询、良恶性评估、 | ||
| 评估报告管理、完成通知的服务。 | ||
| 3 | 数据层 | 包括基因测序仪下机数据和分析结果数据管理(文件对象)、评估数据(PostgreSQL)、用户权限数据(PostgreSQL)等。 |
体系结构设计